Blick in kleinste Zellstrukturen: Krankheitsursachen auf der Spur
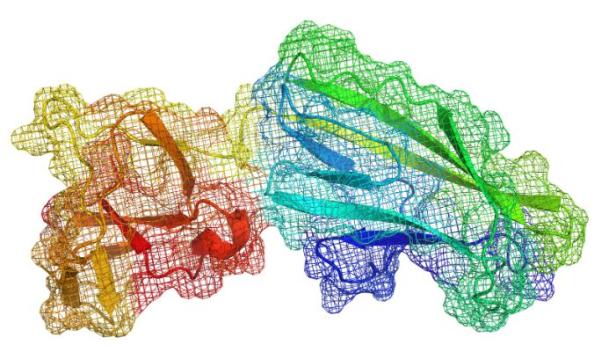
Struktur eines Proteins: Fehler bei den Faltungen können Krankheiten auslösen. Grazer Forscher können noch solche Strukturen mit der Hilfe von künstlicher Intelligenz rascher untersuchen.
Die digitale Revolution durchdringt auch den Gesundheitsbereich - mittels Künstlicher Intelligenz (KI) und Big Data sollen die biomedizinische Forschung vorangetrieben und die Qualität von Diagnosen und Medikamenten verbessert werden. An der Universität Graz wird künstliche Intelligenz zur Erforschung von Proteinstrukturen und molekularen Prozessen eingesetzt. Ihre jüngsten Erkenntnisse aus internationalen Kooperationen haben die Forscher im Journal Science veröffentlicht.
In der biomedizinischen Forschung spielt die Strukturbiologie eine zunehmend größere Rolle. Hier nimmt die Bestimmung von Gestalt und Funktion von Proteinen und der molekulare Prozess der sogenannten Proteinfaltung eine wichtige Position ein. Die Eiweiß-Verbindungen zählen zu den wesentlichen Bausteinen aller Lebensformen – vom Menschen bis hin zu Bakterien sowie Viren. Daneben sind sie für Stoffwechsel, Transportvorgänge und Signalübertragung in allen Zellen verantwortlich. Um diese Funktionen auszuüben, müssen neu hergestellte Proteine in eine spezifische, dreidimensionale Struktur gefaltet werden.
Enscheidende Rolle auch beim Altern
Fehlgefaltete Proteine verklumpen und gelten etwa als eine zentrale Ursache für Erkrankungen wie Alzheimer oder Parkinson. Ebenso spielen Fehler in der Proteinfaltung eine entscheidende Rolle beim Altern. Von Millionen Proteinen ist aber laut den Forschern am Institut für Molekulare Biowissenschaften der Uni Graz nur von einem kleinen Bruchteil der genaue Aufbau bekannt. Die Entschlüsselung mit experimentellen Methoden käme laut Tea Pavkov-Keller einer "Monsteraufgabe" gleich, wie die Forscherin gegenüber der APA schilderte.
Das Problem, mit dem Forscherteams kämpfen, ist, dass sie von vielen Proteinen zwar die Sequenz der Aminosäuren kennen, aus denen sie bestehen, aber nicht die Architektur, also die 3D-Struktur des Proteins. Unter der Leitung der University of Washington in Seattle (USA) hat ein internationales Team, dem auch Forscher aus Graz angehören, eine auf Deep-Learning basierende Methode für Proteinmodellierung entwickelt und getestet. Sie kann Proteinstrukturen und Proteinbindungen mithilfe eines neuronalen Netzwerkes aus einer Aminosäuresequenz vorhersagen. Mithilfe von "RoseTTAFold" konnten die Forschenden an der Uni-Graz zwei bisher unbekannte Proteinstrukturen entschlüsseln.
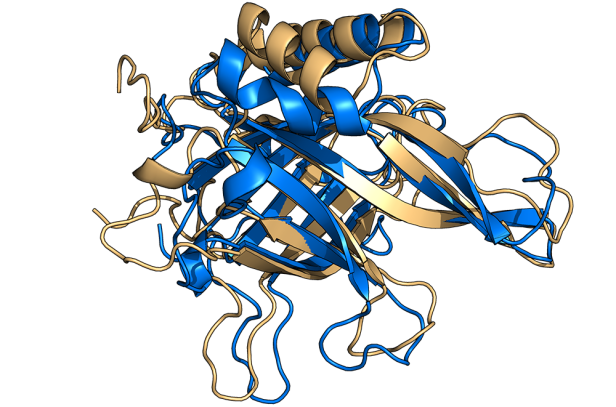
Vorhersage in wenigen Stunden
"Woran ich seit Jahren in Form von Experimenten arbeite, lässt sich nun am Computer in wenigen Stunden mit hoher Präzision vorhersagen und führt in Kombination mit experimentellen Daten zur korrekten Struktur", zeigte sich die Strukturbiologin Pavkov-Keller beeindruckt. Im Falle des einen Proteins, das sie seit Jahren erforscht, ist ihr das mithilfe des neuen Programms innerhalb von zwei Wochen gelungen.
"Zusätzlich können wir vorhersagen, welche Interaktionen zwischen den einzelnen Proteinen passieren. Ebenso erfasst RoseTTAFold jene Eiweiße, die aus mehreren sogenannten Domänen bestehen und sehr oft komplexe Einheiten bilden", erläuterte die Forschungsgruppenleiterin. Das maschinelle Lernen, das auf bekannten Sequenzen und Strukturen aufbaut, macht es darüber hinaus möglich, Funktion und Aufgabe der Proteine zu prognostizieren. Damit werden Verbindungen gezielt veränderbar - etwa wenn es um die Behandlung von Allergien geht.
Drei Grazer Universitäten, die im Forschungsverbund BioTechMed zusammengeschlossen sind, haben ein neues Doktoratsprogramm auf die Beine gestellt. In dem vom Wissenschaftsfonds FWF geförderten Programm "Biomolecular Structures and Interactions" untersuchen junge Forscher Biomoleküle und biomolekulare Komplexe, die eine Rolle bei der Signalübertragung und dem Stoffwechsel von Zellen spielen, wobei der Schwerpunkt auf Proteine, Proteinkomplexen und Lipid/Protein-Wechselwirkungen liegt. "Mit der neuen Anwendung wird auch die Forschung im Doktorandenkolleg beschleunigt", freute sich die Grazer Strukturbiologin.
Kommentare